Inicio / Archivo / Año 6, No 6, septiembre 2023 - agosto 2024 / Paper 18

LA INTELIGENCIA ARTIFICIAL EN EL DESCUBRIMIENTO DE NUEVOS FÁRMACOS PARA EL CONTROL DEL CÁNCER
Juan Manuel Aceves-Hernández*, Alin Miramontes, Jesús Moisés Hernández-Duarte
FES Cuautitlán UNAM
*juanmanuel.is.acevesh@gmail.com
Resumen
El Imatinib, uno de los fármacos que se descubrió in Silico, antes de obtenerse en el laboratorio, se seleccionó para obtener algunos de sus derivados, también in Silico. En este trabajo se presenta la metodología aplicada, así como algunos métodos basados en Inteligencia Artificial que se aplicaron para la predicción de propiedades fisicoquímicas y farmacológicos de los nuevos derivados y su posible obtención en el laboratorio. Basándonos en una descripción de la estructura de una proteína cinasa se pensó en las modificaciones estructurales y de composición al Imatinib, estos derivados se cargaron a dos plataformas que trabajan con redes neuronales para predecir propiedades y actividad biológica, dichas plataformas fueron alimentadas con bases de datos de más de 100,000 compuestos conocidos con y sin actividad biológica. De esta manera fue posible seleccionar tres de los compuestos que presentaron características como potenciales inhibidores de tirosina-cinasas, a los que llamamos compuestos líderes de nuestra búsqueda y que son potenciales fármacos para control de cáncer, como el Imatinib.
Palabras clave: In Silico, modelado molecular, derivados Imatinib, Apoptosis, redes Neuronales.
Introducción
El cáncer es un proceso de crecimiento y diseminación incontrolado de células y puede aparecer prácticamente en cualquier lugar del cuerpo, el tumor suele invadir el tejido circundante y puede provocar metástasis en puntos distantes del organismo, es causado por alteraciones genéticas y epigenéticas, provocadas por factores hereditarios y ambientales. El cáncer es una de las principales causas de morbilidad y la segunda causa de muerte en el mundo, casi 1 de cada 6 defunciones en el mundo se debe a esta enfermedad (Organización Mundial de la Salud, 2022). La leucemia es un cáncer en las células sanguíneas, las cuales en su mayoría se forman en la médula ósea, estas células no funcionan adecuadamente y desplazan las células sanguíneas normales; existen diversos tipos de leucemia y estos dependen del tipo de célula sanguínea que se convierte en cancerígena. La leucemia se presenta más frecuentemente en adultos mayores a los 55 años, pero también es el cáncer más común en menores de 15 años (Instituto Nacional de Cancerología, 2017). La leucemia es responsable aproximadamente del 3% de todos los cánceres humanos, con una incidencia mundial de aproximadamente 6 a 10 casos por 100,000 personas. En específico, la Leucemia Mieloide Crónica (LMC) tiene una incidencia de 1-2 casos por cada 100,000 personas y es responsable del 15-20% de todas las leucemias del adulto.
La Leucemia Mieloide Crónica (LMC) fue el primer cáncer vinculado casualmente a una anomalía genética, siendo un cromosoma anormal el que se asociaba a esta enfermedad, denominado “Cromosoma Philadelphia” (Ph1), esto consiste en una mutación BCR-ABL (oncoproteína) llamado así, ya que existe una translocación o intercambio de material genético entre los brazos largos de los cromosomas 9 y 22 (Catalina, 2010); se ha comprobado que este cromosoma está presente en el 95% de los casos de LMC y que la transformación se origina en la célula madre hematopoyética primitiva, lo que lleva a una proliferación clonal de células madre mieloides malignas y con ello un número excesivo de células mieloides en todas las fases de maduración (Carretero, 2003).
A nivel molecular, el Ph1 se expresa en una proteína anormal de la familia de las cinasas llamada tirosina-cinasa BCR-ABL (dependiendo de la región donde ocurra el re-arreglo genómico, puede dar origen a varios tipos de proteínas de fusión BCR-ABL, siendo las más comunes las de 210, 190 y 230 kDa, cuya actividad anormal afecta procesos básicos de la célula tales como señalización mitogénica constitutivamente activa, adherencia defectuosa a células estromales y matriz extracelular así como una reducción en la respuesta a estímulos inductores de apoptosis, es decir que tales células no pueden llevar a cabo sus funciones de manera adecuada y que además, no mueren en un ciclo normal, sino que se multiplican hasta desplazar a las células sanas, comprometiendo el sistema inmune del paciente (Camargo, 2008).
El imatinib es el primer inhibidor de la tirosina quinasa BCR-ABL disponible para uso clínico, es un derivado de la 2-fenilaminopirimidina (un antagonista de las tirosinquinasas proteínicas y ejerce una inhibición potente y específica de BCR-ABL; el imatinib actúa específicamente bloqueando el lugar de unión para el ATP en la tirosina quinasa BCR-ABL, lo que resulta en impedir la transducción de señales necesarias para la proliferación celular e inducir la apoptosis en las células Ph+ y que albergan tirosina quinasa BCR-ABL, es decir, inhibe sólo la causa específica de la LMC siendo un tratamiento eficaz y selectivo que cumple con el objetivo del tratamiento dirigido a la causa molecular (Carretero, 2003).
Se sabe que los costos de un fármaco desarrollado y aprobado por una compañía farmacéutica es en promedio de 1 a 2 mil millones de dólares y puede durar ente 10-17 años (Maia et al., 2020); esto es bajo el método histórico de prueba y error que no resulta eficiente en la actualidad; sin embargo, el diseño racional de fármaco se basa en el conocimiento de las rutas bioquímicas involucradas en la enfermedad o procesos biológicos de interés, hoy en día los métodos computacionales para este fin han contribuido al análisis eficiente de datos, una mejor y reducida selección de moléculas para evaluación experimental y a la formulación de hipótesis sobre los mecanismos de acción, así como al diseño de nuevas estructuras químicas más selectivas y específicas, lo que resulta en terapias más seguras y accesibles para el paciente, a menor costo y en menor tiempo (Saldívar, 2016).
La inteligencia artificial (IA) se está utilizando cada vez más en el diseño de fármacos para acelerar el proceso de descubrimiento de nuevos medicamentos y reducir el costo y el riesgo asociados con los ensayos clínicos. La IA puede ayudar a los científicos a predecir la actividad biológica de las moléculas y optimizar las propiedades farmacocinéticas, como la absorción, la distribución, el metabolismo y la eliminación. Además, puede ayudar a los investigadores a identificar posibles efectos secundarios y a predecir la toxicidad de los compuestos (Baum et al., 2021). En la Figura 1, se presentan las aplicaciones de la inteligencia artificial en la búsqueda de fármacos.
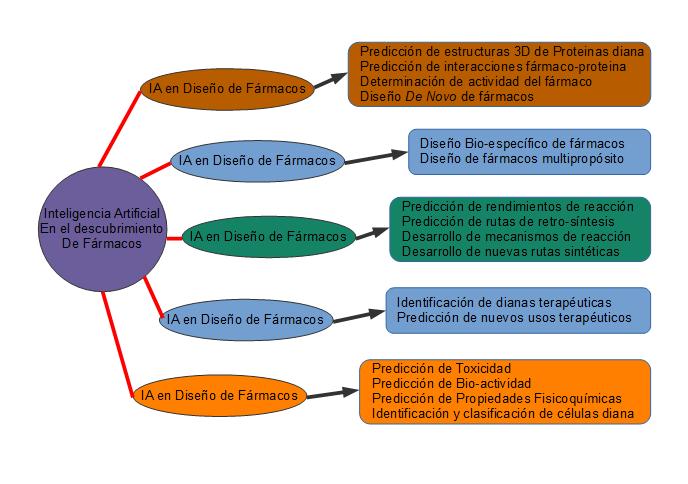
Figura 1. Adaptado de Debleena et al. (2021).
Objetivo
El principal objetivo de este trabajo es usar las herramientas de bio-informática, redes neuronales y de Inteligencia Artificial, para obtener tres derivados de Imatinib con posibles usos en el tratamiento de cáncer, para luego prepararlos en el laboratorio y probar su potencial efecto biológico en líneas celulares cancerosas.
Otro objetivo es mostrar las dificultades y retos en el uso de esta metodología en la predicción de estructuras 3D en proteínas y estructuras químicas. Finalmente, otro objetivo es mostrar las grandes oportunidades y usos que ofrece la Inteligencia Artificial en el descubrimiento de nuevos fármacos y el uso de los ya descubiertos para otros usos terapéuticos y otras enfermedades. En la Tabla 1 se presentan las herramientas usadas en el descubrimiento de fármacos.
Tabla 1. Ejemplos de herramientas de IA que se usan en el descubrimiento de fármacos.
| Herramientas | Detalles | Website URL |
|---|---|---|
| DeepChem | Modelo MLP que usa un sistema de AI basado en Python para encontrar un candidato adecuado en la búsqueda de fármacos | https://github.com/deepchem/deepchem |
| DeepTox | Software que predice la toxicidad de un total de 12000 fármacos | http://www.bioinf.jku.at/research/DeepTox/ |
| DeepNeuralNetQSAR | Sistema basado en Python impulsado por herramientas computacionales que ayudan a la detección de la actividad molecular de compuestos | https://github.com/Merck/DeepNeuralNet-QSAR |
| ORGANIC | Una herramienta de generación molecular que ayuda a crear moléculas con las propiedades deseadas | https://github.com/aspuruguzik-group/ORGANIC |
| PotentialNet | Utiliza NN para predecir la afinidad de unión de ligandos | https://pubs.acs.org/doi/full/10.1021/acscentsci.8b00507 |
| Hit Dexter | Técnica de ML para predecir moléculas que podrían responder a ensayos bioquímicos | http://hitdexter2.zbh.unihamburg.de |
| DeltaVina | Una función de puntuación para restablecer la afinidad de unión entre fármaco y ligando | https://github.com/chengwang88/deltavina |
| Neural graph fingerprint | Ayuda a predecir propiedades de nuevas moléculas Predice estructuras 3D de proteínas | https://github.com/HIPS/neural-fingerprint |
| AlphaFold | Predice estructuras 3D de proteínas | https://deepmind.com/blog/alphafold |
| Chemputer | Ayuda a informar el procedimiento de síntesis química en formato estandarizado | https://zenodo.org/record/1481731 |
Adaptado de Debleena et al. (2021).
Materiales y métodos
Para la selección de la proteína de referencia se utilizó la base de datos Protein Data Bank (PDB), considerando proteínas del tipo Tirosina Cinasa expresada en Homo sapiens. Los criterios de selección fueron la resolución ≤2.5 Å y que el método fuera Resonancia Magnética Nuclear (RMN) o Difracción de Rayos-X. La proteína seleccionada fue la 3GVU que pertenece al gen ABL, ABL2.
Esta estructura se cargó a la plataforma CavityPlus 2 (LINK). En la cual se corrió análisis para detección de cavidades potenciales para interacción con fármacos, las cavidades con alta probabilidad de interacción con un fármaco se analizaron con el submódulo CavPharmer, el cual trabaja bajo un modelado de grupos farmacóforos basados en el receptor (proteína).
Se hizo una propuesta de 11 modificaciones al Imatinib, las estructuras planas se generaron a través del programa ChemSketch, obteniendo su código SMILES. El código SMILES fue utilizado en la plataforma Molinspiration en la cual se solicitó predicción de propiedades fisicoquímicas y actividad biológica. El SMILES también se utilizó para el análisis en el programa Osiris, en el cual se obtuvieron tanto descriptores como toxicología. Con base en esa información se seleccionaron las moléculas con un coeficiente de inhibición de Tirosina-cinasa mayor al que presenta IMATINIB.
Para la optimización de las moléculas de Imatinib y de las moléculas lideres, se empleó el paquete de software Gaussian 16, que está en la Supercomputadora Miztli de la UNAM, previamente se usó el programa Spartan y Gaussview para encontrar el confórmero de menor energía.
Resultados
En la Figura 2, se presenta la estructura no optimizada del imatinib
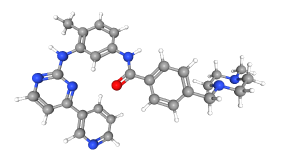
Figura 2. Estructura Imatinib en Pubchem.
En la Figura 3, se presenta la conformación en 3D, de la proteína 3GVU, en la que se hizo el Docking con cada uno de los ligandos líderes. Mientras que en la Figura 4, se presentan las Cavidades de la proteína 3GVU identificadas por CavityPlus2
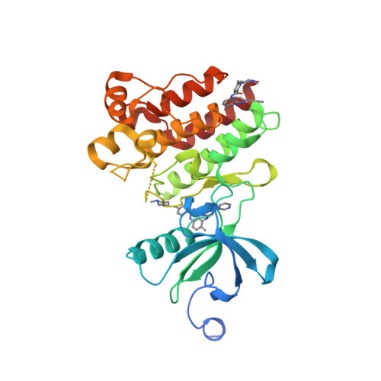
Figura 3. Proteína 3GVU en PDB.
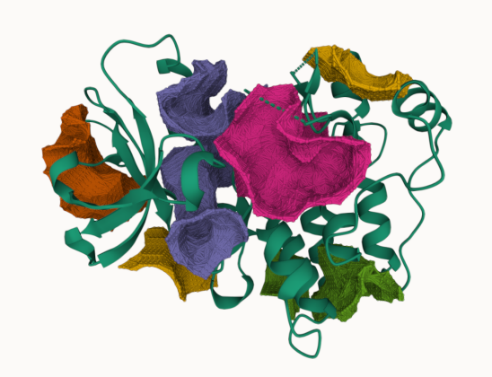
Figura 4. Cavidades de la proteína 3GVU identificadas por CavityPlus2.
En la Tabla 2, se presentan los descriptores de las cavidades en la proteína 3GVU.
Tabla 2. Descriptores de las cavidades identificadas por CavityPlus.
| # | Estimado Máximo pKd | Promedio Estimado pKd | Puntaje del Fármaco | Facilidad para convertirse en Fármaco |
|---|---|---|---|---|
| 1 | 10.54 | 6.94 | 268 | Medio |
| 2 | 11.76 | 6.82 | 1863 | Fuerte |
| 3 | 11.62 | 6.6 | -303 | Débil |
| 4 | 11.03 | 6.4 | 4 | Medio |
| 5 | 9.44 | 5.85 | -749 | Débil |
| 6 | 9.24 | 5.79 | 992 | Fuerte |
| 7 | 8.63 | 5.58 | -567 | Débil |
| 8 | 8.37 | 5.49 | -949 | Débil |
En las Figuras 5, 6 y 7 se presentan las estructuras optimizadas de las moléculas 2, 6 y 11, respectivamente, que arrojaron las mejores características farmacológicas, para ser usados como potenciales fármacos contra el cáncer, faltando sólo hacer pruebas en diferentes líneas celulares cancerosas.
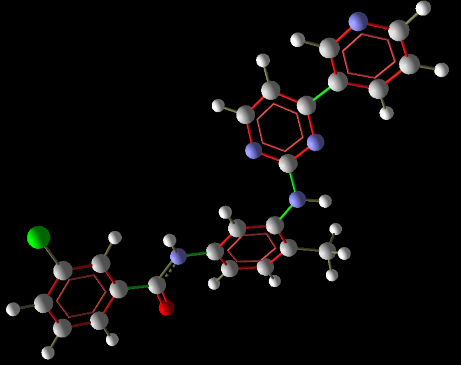
Figura 5. Estructura optimizada de “Molécula 2”.
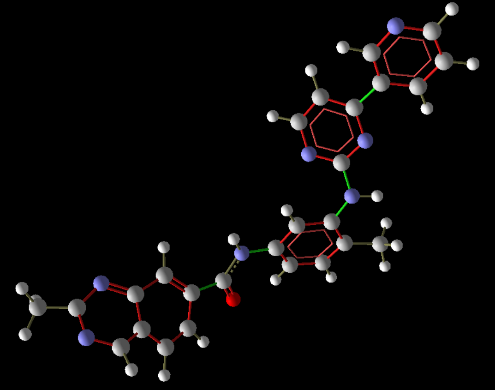
Figura 6. Estructura optimizada de “Molécula 6”.
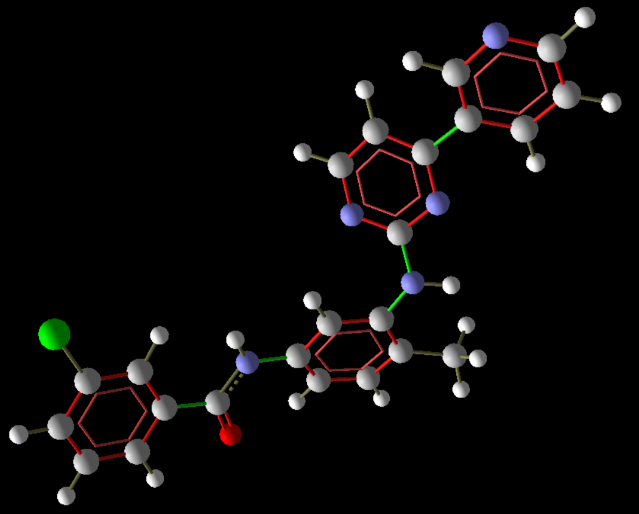
Figura 7. Estructura optimizada de “Molécula 11”.
Discusión
Una vez que se obtuvieron los valores de los diferentes descriptores, se encontró que la molécula 2 presentaba mejores características que la molécula 6 y esta, era mejor que la molécula 11, pero se tienen que validar estos resultados con las pruebas in vitro, a realizarse en líneas celulares cancerosas, ya que puede ser que cada molécula presente una acción terapéutica mejor en diferentes líneas celulares.
Conclusión
De los resultados obtenidos, se puede concluir lo siguiente: 1) que el uso de la Inteligencia Artificial y de redes neuronales es muy útil en el descubrimiento de nuevos fármacos, ya que se ahorra tiempo, recursos económicos y esfuerzo. 2) que una vez que se encuentra un ligando líder in silico, se puede iniciar el trabajo de síntesis de dicho compuesto y así acelerar el trabajo de investigación en líneas celulares y en las pruebas clínicas. 3) que a pesar de las ventajas que presenta esta metodología, aún se presentan dificultades en la obtención de parámetros que aseguren el éxito de la investigación y de óptimos resultados. El trabajo subsecuente es la obtención en el laboratorio de las moléculas 2, 6 y 11, para hacer pruebas clínicas.
Referencias
Baum, Z. J., Yu, X., Ayala, P. Y., Zhao, Y., Watkins, S. P., & Zhou., Q. (2021). Artificial Intelligence in Chemistry: Current Trends and future directions. Journal of Chemical Information and Modeling, 3197-3212.
Bruton, J. L. (2007). Goodman & Gilman: Las bases farmacológicas de la terapéutica (11 ed.). (J. O.-T. José Rafael Blengio Pinto, Trans.) Colombia: McGRAW-HILL.
Camargo, D. S. M. (2008, octubre-diciembre). Tratamiento con Imatinib y farmacogenotipo CYP3A4 en relación con la expansión clonal Ph(+) en leucemia mieloide crónica (LMC). Colombia Médica, 39(4), 314-322.
Carretero, M. (2003, enero). Imatinib. Actualidad Científica: Medicamentos de vanguardia, 22(1), 138-140.
Catalina, M. V. T. (2010, enero-junio). Leucemia mieloide crónica: diagnóstico y tratamiento. CES Médica, 24(1), 97-108.
D. Van Der Spoel, E. L. (2005). GROMACS: fast, flexible and free. Journal of Computacional Chemistry, 26, 1701-1718.
Debleena, P., Gaurav, S., Snehal, S., Dnyaneshwar, K., Kiran, K., & Rakesh. K. T. 2021. Artificial intelligence in drug discovery and development, Drug Discovery Today, 26(1), 80-93.
Saldívar, G. F. D. (2016). Descubrimiento y desarrollo de fármacos: un enfoque computacional. Educación Química, 28, 51-58.
Hall, J. E. (2016). Guyton y Hall: Tratado de fisiología médica. Elsevier.
Maia, E. h., Assis, C. L., Oliveira, T. A., Silva, A. M., & Taranto, A. G. (2020). Structure-Based virtual Screening: From classical to Artificial Intelligence. Frontiers in Chemistry.
Murray, R. (2013). HARPER: Bioquímica. España: McGRAW-HILL.
Sexto Congreso Nacional de Tecnología 18, 19 y 20 de octubre de 2023,
celebrado en formato virtual

D. R. © UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO.
Excepto donde se indique lo contrario esta obra está bajo una licencia Creative Commons Atribución No comercial, No derivada, 4.0 Internacional (CC BY NC ND 4.0 INTERNACIONAL).
https://creativecommons.org/licenses/by-nc-nd/4.0/deed.es

ENTIDAD EDITORA
Facultad de Estudios Superiores Cuautitlán.
Av. Universidad 3000, Universidad Nacional Autónoma de México, C.U., Delegación Coyoacán, C.P. 04510, Ciudad de México.
FORMA SUGERIDA DE CITAR:
Aceves-Hernández, J. M., Miramontes, A., y Hernández-Duarte, J. M. (2023). La inteligencia artificial en el descubrimiento de nuevos fármacos para el control del cáncer. MEMORIAS DEL CONGRESO NACIONAL DE TECNOLOGÍA (CONATEC), Año 6, No. 6, septiembre 2023 - agosto 2024. Facultad de Estudios Superiores Cuautitlán. UNAM. https://tecnicosacademicos.cuautitlan.unam.mx/CongresoTA/memorias2023/Mem2023_Paper18-E.html