Inicio / Archivo / Año 7, No 7, septiembre 2024 - agosto 2025 / Paper 02

CONFECCIÓN DE UN VECTOR PARA EL SILENCIAMIENTO DEL GEN LcACR2 DE Lupinus campestris
Luciano Antonio Juárez-Díaz1, César Díaz-Pérez2, Francisco Javier Hernández-Flores1, Melina del Real-Monroy1 y Lenin Sánchez-Calderón1*
1Universidad Autónoma de Zacatecas (UAZ), Unidad Académica de Ciencias Biológicas. Campus II UAZ y 2Departamento de Ingeniería Agroindustrial, Facultad de Agrobiología, Campus Salvatierra, Universidad de Guanajuato, Guanajuato, México
*xamachana22@gmail.com
Resumen
La toxicidad generada por el arsénico (As) altera procesos fisiológicos que pueden llevar a la muerte a las plantas. No obstante, éstas han desarrollado mecanismos de resistencia al As, como la detoxificación mediada por la enzima arsenato reductasa 2 (ACR2), que reduce el As(V) a As(III), y facilita su acumulación en la vacuola o la exclusión del citoplasma. Esta enzima ha sido identificada en especies de plantas como Pteris vittata, Arabidopsis thaliana y Oryza sativa. Lupinus campestris ha mostrado resistencia in vitro a As en concentraciones de hasta 100 µM, acumulándolo en sus raíces sin mostrar signos de toxicidad. El uso de vectores específicamente diseñados para expresar hpRNAs para el silenciamiento génico es útil para generar herramientas génicas que ayuden en los procesos de biorremediación. El objetivo de este trabajo fue clonar un vector codificante de hpRNA para el silenciamiento postranscripcional del gen LcACR2 en L. campestris por lo que esta planta, podrá translocar el As al sistema de brotes haciéndola una planta eficiente para la fitoextracción de As. Dado que no se cuenta con el genoma de L. campestris, se planteó usar secuencias de genes del género Lupinus en el diseño de oligonucleótidos para amplificar el gen LcACR2. Para este fin, se construyó un árbol filogenético con secuencias de ACR2 de diversas especies y se buscaron las regiones conservadas en la familia ACR2. Con los oligonucleótidos diseñados, se amplificó una región de 350 pb del gen LcACR2 de L. campestris y se clonó en un vector para silenciarlo.
Palabras clave: ACR2 (arsenato reductasa 2), silenciamiento génico, fitorremediación.
Introducción
El arsénico (As) es un metal pesado que en su forma química arseniato [As(V)], puede ser tomado por los transportadores PHT1 (Phosphate Transporter 1) debido a su similitud química con el fosfato. Dentro de la célula, el arsénico es tóxico, ya que puede modificar sitios estructurales y catalíticos de las proteínas y genera especies reactivas de oxígeno, lo que termina por alterar procesos fisiológicos, como la fotosíntesis y la respiración celular. Uno de los mecanismos de detoxificación de As en el citoplasma, consiste en la reducción del As(V) a As(III), mediante la enzima As(V) reductasa (ACR2), el As(III) forma complejos con las proteínas fitoquelatinas y es acumulado en las vacuolas. La enzima ACR2 fue inicialmente aislada en bacterias y levaduras, y posteriormente se ha identificado en plantas como Arabidopsis thaliana, Pteris vittata y Oryza sativa. De acuerdo con lo reportado, la enzima ACR2 conserva un motivo HC(X5)R que forma parte del sitio activo, además, por su dominio rodanasa, y está relacionada con las tirosin-fosfatasas CDC25 que participan en el ciclo celular. Para el mecanismo de reducción la actividad de AsV está dada por un grupo de cisteínas: Cys-134, Cys-136, Cys-141 y Cys-145 (Cesaro et al., 2015; Ghori et al., 2019).
Clonación y silenciamiento génico
La clonación es una técnica molecular donde se inserta uno o más fragmentos de DNA en un vector que puede ser usado para diferentes fines. Este proceso incluye obtener el DNA de interés, ligarlo a un vector, transformar una bacteria huésped y seleccionar a aquellas transformadas y purificar las múltiples copias del vector que contiene el fragmento de interés. Los vectores más comunes son plásmidos, aunque también se utilizan bacteriófagos y cósmidos. El sistema de clonación Gateway, basado en el fago λ, es ampliamente usado por su versatilidad en múltiples aplicaciones entre ellas el silenciamiento génico (Ashwini et al., 2016; Bertero et al., 2017; Reece y Walhout, 2018). El silenciamiento post-transcripcional (PTSG), utiliza estructuras de ARN tallo-asa para degradar el ARN mensajero blanco, impidiendo su traducción. Este proceso es útil para estudiar la función de un gen particular (Ashfaq et al., 2020; Brodersen y Voinnet, 2006).
Lupinus campestris
Lupinus campestris es una especie que se distribuye en suelos contaminados con metales pesados en Zacatecas, México. En medios de cultivo MS 0.5X adicionados con 100 µM de As(III o V), plántulas de L. campestris acumularon As en la raíz sin mostrar signos de toxicidad. Se observó que el As no se translocó a la parte aérea, lo cual hace que la planta no sea apta para la fitoextracción de As. Lo anterior sugiere que esta especie puede estar detoxificando el As(V), mediante una enzima similar a la ACR2 para su posterior acumulación en la raíz.
Objetivo
Dado lo anterior, este trabajo tuvo como objetivo identificar a un homólogo de la proteína ACR2 en L. campestris (LcACR2) y construir un vector codificante de hpRNA para inducir el silenciamiento génico de LcACR2 en L. campestris, con lo cual se puede generar una variedad que podrá translocar el As al sistema de brotes haciéndola una planta eficiente para la fitoextracción de As.
Materiales y métodos
Análisis bioinformático
Para identificar las proteínas homólogas a ACR2 (AtACR2; número de acceso: NP_568119) de A. thaliana en especies de Lupinus, se llevó a cabo una búsqueda con BLASTn. Se seleccionaron las secuencias con una cobertura de secuencia mayor a 80%. Las secuencias obtenidas se alinearon mediante ClustaIW en Bioedti 7.2 (Hall, 1999). Se hizo una reconstrucción de historia evolutiva usando el método de mínima evolución con el programa MEGA 7 (Kumar et al., 2016). Para la predicción funcional de la enzima LaACR2, se llevó a cabo un análisis de dominios mediante el servidor CDD (Wang et al., 2022).
Material biológico y transformación
El fragmento del gen LcACR2 fue obtenido a partir de cDNA del tejido de plántulas de L. campestris que crecieron en medio de cultivo MS 0.5X adicionado con 100 µM de arsenato de potasio [As(V)]. El cDNA se obtuvo usando el kit SuperScript ® First-Strand Synthesis System for RT-PCR de Invitrogen ™, usando los oligos(dT), de acuerdo con las instrucciones del proveedor. Para la clonación se siguieron los protocolos del kit BP y LR clonase, usado al vector donador PDONR221 (ThermoFisher Scientific) con el marcador de selección kanamicina, y como vector destino se utilizó el plásmido pHELLSGATE12 que otorga resistencia a espectinomicina. Para la transformación se utilizaron las cepas de E. coli One Shot OmniMAX 2 T1R (ThermoFisher Scientific) químicamente competente y DB3.1:pHELLSGATE12.
Resultados
Reconstrucción filogenética
Con el fin de encontrar secuencias homólogas de la enzima AtACR2 de A. thaliana en especies de Lupinus, se llevó a cabo la reconstrucción filogenética de esta familia de proteínas. En este análisis las secuencias proteicas de Lupinus albus y Lupinus angustifolius se agruparon con un soporte del 100% en los valores de Bootstrap (Figura 1).
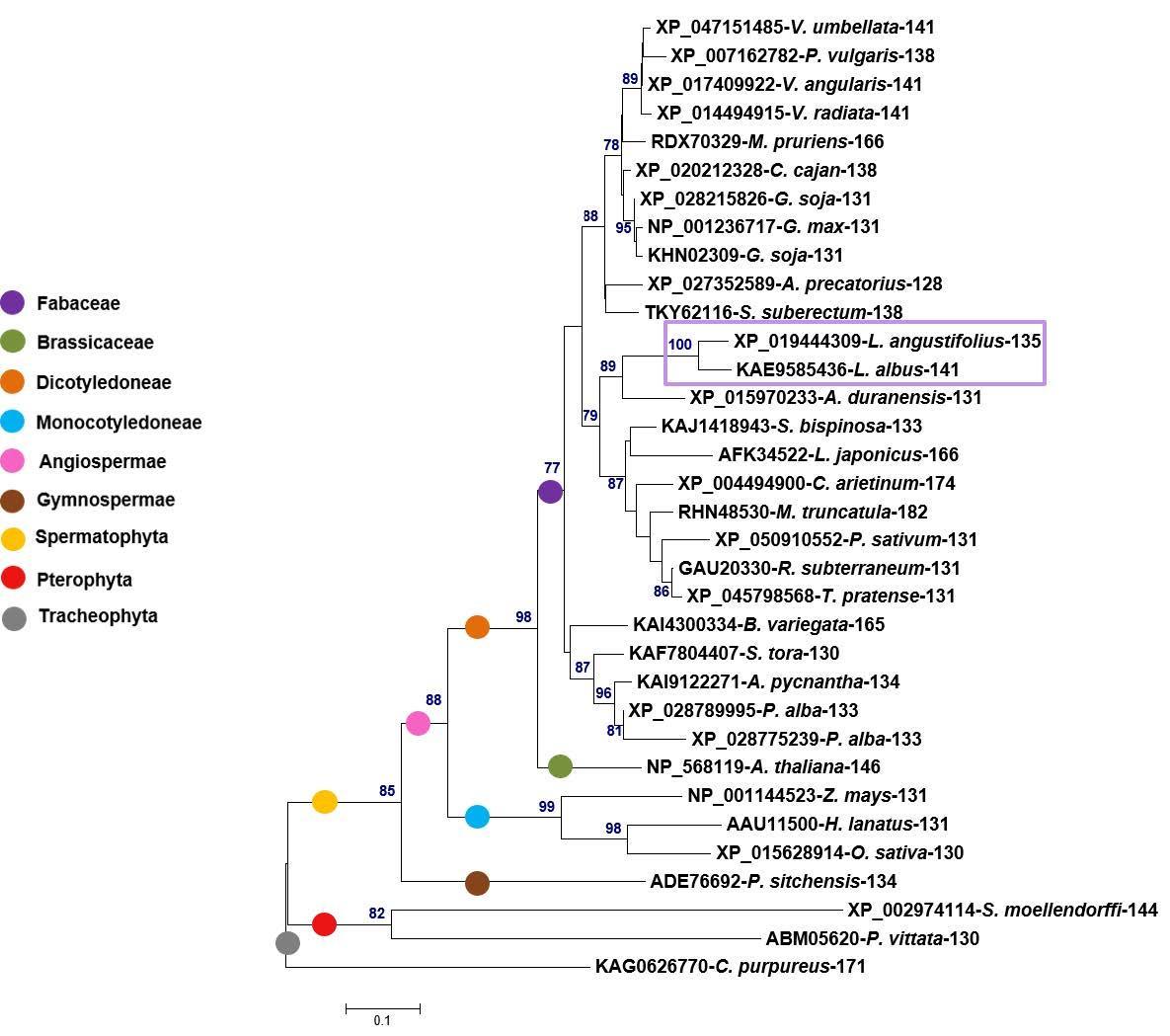
Figura 1. Reconstrucción filogenética mediante ME de proteínas similares a AtACR2 dentro de Tracheophyta. Se muestran veintiséis secuencias de la familia Fabaceae. El árbol filogenético se reconstruyó mediante el método de Mínima evolución. Se muestran los valores Bootsrap mayores a 70.
Predicción de función de proteínas y deducción de regiones funcionales
Para encontrar las regiones conservadas de la familia, importantes para la función enzimática, se analizaron las proteínas encontradas en el servidor CDD. Se observó que todas las proteínas tienen dos dominios conservados (Figura 2), el dominio Acr2p (cd01531) , propio de las As(V) reductasas, que abarcan aproximadamente 110 residuos, y el dominio de rodanasa (cl00125) que está formado de alrededor de 100 residuos. Se observó que un motivo consenso HC(X5)R se encuentra conservado en todas las secuencias de la familia Fabaceae la cual forma parte del sitio activo de las enzimas As(V) reductasas (2). Las proteínas del género Lupinus conservan los aminoácidos característicos del sitio activo de la familia ACR2, que está formado por un núcleo de cisteínas (His-41, C-122, C-124 y C-129). Con esta información se diseñaron los oligonucleótidos que flanquean la región conservada que contiene el dominio funcional Acr2p.
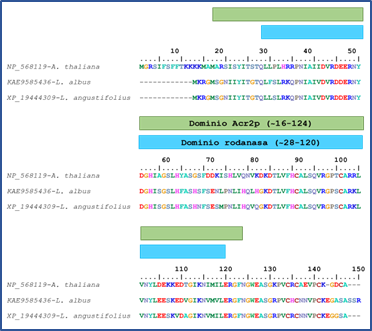
Figura 2. Dominios conservados en la familia ACR2. Se muestran las secuencias de las proteínas de A. thaliana (NP_568119), L. albus (KAE9585436) y L. angustifolius (XP_019444309). Se muestran en verde y azul los dominios Acr2p y rodanasa respectivamente.
Obtención de un vector de silenciamiento para el gen LcACR2
Mediante una PCR touchdown se amplificó un fragmento de 350 pb gen LcACR2 a partir de cDNA obtenido de tejido plántulas de L. campestris crecido en presencia de As(V) (Figura 3 A). Mediante la reacción BP, este fragmento se clonó en el vector de entrada pDONR221 (Figura 3 B) y se subclonó en el vector de destino pHELLSGATE12 usando la reacción LR (Figura 3 C).
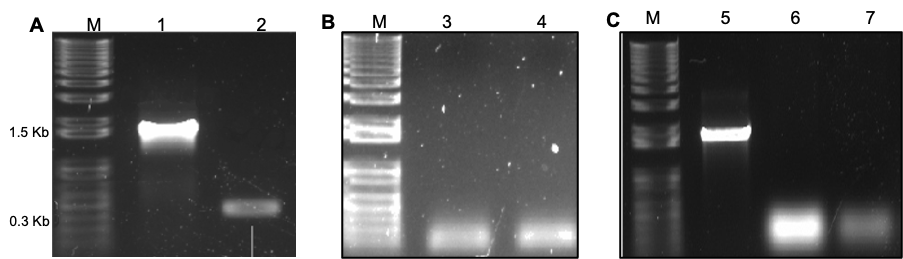
Figura 3. Clonación del fragmento del gen LcACR2. A) Amplificación del gen LcACR2 (fragmento de 350 pb del gen LcACR2) a partir del cDNA de plantas de 6 días de crecimiento en MS 0.5X adicionado con 100 µM de As(V). B) Amplificación del fragmento del gen del gen LcACR2 de clonas positivas de la reacción BP. C) Amplificación del fragmento del gen del gen LcACR2 de clonas positivas de la reacción LR. M.- marcador de tamaño molecular; 1 y 5.- control positivo (amplificación del gen PHT1); 2.- amplificación del fragmento de LcACR; 3 y 4.- clonas positivas de la reacción BP; 6 y 7.- clonas positivas de la reacción LR.
Después de la reacción de recombinación en este vector, el fragmento del gen LcACR2 se insertó en sentido y en antisentido flanqueando un intrón, lo cual permitirá la formación del tallo asa para el silenciamiento del gen LcACR2.
Discusión
Las proteínas de la familia ACR2 están involucradas en la detoxificación del As en S. cerevisiae y en varias especies vegetales como A. thaliana y P. vittata. Los análisis filogenéticos, la similitud de secuencia y la conservación de motivos funcionales sugiere que las proteínas de la familia podrían conservar la función enzimática, incluso entre distintos grupos taxonómicos (Wolko et al., 2011). Dado que el genoma de L. campestris no ha sido secuenciado aún, se utilizaron las secuencias de dos especies del género Lupinus, L. albus y L. angustifolius, para la búsqueda de proteínas similares a ACR2 de A. thaliana.
En la reconstrucción filogenética generada en esté trabajo (Figura 1), fue posible recuperar varias secuencias del clado Tracheophyta, mostrando que es posible encontrar proteínas ACR2 desde los clados basales como Pteridophyta s.l. hasta aquellos de más reciente aparición como Angiospermae. Esto sugiere que los genes que codifican a las proteínas ACR podrían compartir un origen común, y que por lo tanto los genes ACR2 de cada una de estas especies son ortólogos, como se ha sugerido en otros trabajos (Dhanker et al., 2006).
Las proteínas encontradas contienen los dominios conservado Acr2p y rodanasa. Lo anterior también se relaciona con la tirosina fosfatasa CDC25 (Bordo y Bork 2002) pues contienen un dominio rodanasa lo que sugiere la función de reductasa. Se observó un motivo conservado HC(X5)R, que se encuentra en todas las secuencias de la familia Fabaceae el cual forma parte del sitio activo de las enzimas As(V) reductasas. En el dominio de rodanasa (cl00125) de las secuencias del género Lupinis se encontraron los residuos que son parte del sitio catalítico reportado para la proteína ACR2 (Bordo & Bork, 2002), lo que sugiere que las enzimas pueden ser funcionales.
Dado que se ha construido un vector codificante de hpRNA para el silenciamiento génico de ACR2 en L. campestris, este puede ser de gran utilidad para, además de corroborar in vitro su función, aumentar la translocación de As a la parte aérea, al silenciar el gen ACR2 en L. campestris.
Conclusión
Se encontraron miembros de la familia ACR2 en el clado Fabaceae, específicamente dos proteínas dentro del género Lupinus. Estas proteínas contienen los dominios conservados Acr2p (cd01531) y rodanasa (cl00125), además del motivo HC(X5)R y los residuos importantes del sitio activo H-41, C-122, C-124 y C-129, por lo que es probable que conserven la función AsV-reductasa. Se logró amplificar un fragmento del gen LcACR2, por los que se concluye que existe un homólogo de la familia ACR2 en L. campestris. Se logró la generación de un vector para el silenciamiento del gen LcACR2 de L. campestris.
Agradecimientos
Este trabajo fue posible con los fondos provenientes de los proyectos: Convocatoria Institucional de Investigación Científica 2024 de la Universidad de Guanajuato, proyecto 064/2024. Ciencia Básica CB-2010-01 000000000156851 y UAZ-202138539. A la Dra. Gloria Guerrero Manríquez del Laboratorio de Bioquímica Molecular e Inmunología, y a la Dra. Rosa María Ramírez Santoyo y la Dra. Luz Elena Vidales Rodríguez del Laboratorio de Biología de Bacterias y Hongos por permitir el uso de sus equipos.
Referencias
- Ashfaq, M.A., Dinesh, K.V., Soma, S.R.P., Anil, K.C., Sai, K.K., Narasimha, R.N., Tarakeswari, M., Sujatha, M. (2020). Post-transcriptional gene silencing: Basic concepts and applications. Journal of biosciences, 45(1): 128.
- Ashwini, M., Murugan, S.B., Balamurugan, S., Sathishkumar, R. (2016). Advances in molecular cloning. Molecular Biology, 50: 1-6.
- Bertero, A., Brown, S., Vallier, L. (2017). Methods of cloning. In Basic science methods for clinical researchers. 19-39. Elsevier.
- Bordo, D., Bork, P. (2002). The rhodanese/Cdc25 phosphatase superfamily. Sequence-structure-function relations. EMBO Reports 3(8): 741-746. DOI: 10.1093/embo-reports/kvf150
- Brodersen, P., Voinnet, O. (2006). The diversity of RNA silencing pathways in plants. TRENDS in Genetics, 22(5): 268-280.
- Cesaro, P., Cattaneo, C., Bona, E., Berta, G., Cavaletto, M. (2015). The arsenic hyperaccumulating Pteris vittata expresses two arsenate reductases. Scientific Reports, 5(1): 14525.
- Dhankher, O.P., Rosen, B.P., McKinney, E.C., Meagher, R.B. (2006). Hyperaccumulation of arsenic in the shoots of Arabidopsis silenced for arsenate reductase (ACR2). Proceedings of the National Academy of Sciences, 103(14), 5413-5418.
- Ghori, N.H., Ghori, T., Hayat, M., Imadi, S., Gul, A., Altay, V., Ozturk, M. (2019). Heavy metal stress and responses in plants. International journal of environmental science and technology, 16: 1807-1828.
- Hall, T.A. (1999). BioEdit: A User-Friendly Biological Sequence Alignment Editor and Analysis Program for Windows 95/98/NT. Nucleic Acids Symposium Series, 45: 95-98.
- Kumar, S., Stecher, G., Tamura, K. (2016). MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets. Mol Biol Evol, 33(7): 1870-1874.
- Reece, H.J.S., Walhout, A.J. (2018). Gateway recombinational cloning. Cold Spring Harbor Protocols, 2018(1), pdb. top094912.
- Wang, J., Chitsaz, F., Derbyshire, M.K., Gonzales, N.R., Gwadz, M., Lu, S., Marchler, G.H., Song, J.S., Thanki, N., Yamashita, R.A., Yang, M., Zhang, D., Zhen, C., Lanczycki, C.J., Marchler, B.A. (2022). The conserved domain database in 2023. Nucleic Acids Research, 51(D1), D384-D388.
- Wolko, B., Clemens, J.C., Naganowska, B., Nelson, M.N., Yang, H. (2011). Lupinus. Capítulo 9. En: Chittaranjan Kole (editor) Wild Crop Relatives: Genomic and Breeding Resources. Springer-Verlag Berlin Heidelberg. 153-206. DOI 10.1007/978-3-642-14387-8.
Séptimo Congreso Nacional de Tecnología 16,
17 y 18 de octubre de 2024,
celebrado en formato virtual

D. R. © UNIVERSIDAD NACIONAL AUTÓNOMA DE MÉXICO
Excepto donde se indique lo contrario esta obra está bajo una licencia Creative Commons
Atribución No comercial, No derivada, 4.0 Internacional (CC BY NC ND 4.0
INTERNACIONAL).
https://creativecommons.org/licenses/by-nc-nd/4.0/deed.es

ENTIDAD EDITORA
Facultad de Estudios Superiores Cuautitlán.
Av. Universidad 3000, Universidad Nacional Autónoma de México, C.U., Delegación Coyoacán, C.P. 04510, Ciudad de México.
FORMA SUGERIDA DE CITAR:
Juárez-Díaz, L. A., Díaz-Pérez, C., Hernández-Flores, F. J., del Real-Monroy, M., y Sánchez-Calderón, L. (2024). CONFECCIÓN DE UN VECTOR PARA EL SILENCIAMIENTO DEL GENLcACR2 DE Lupinus campestris. MEMORIAS DEL CONGRESO NACIONAL DE TECNOLOGÍA (CONATEC), Año 7, No. 7, septiembre 2024 - agosto 2025. Facultad de Estudios Superiores Cuautitlán. UNAM https://tecnicosacademicos.cuautitlan.unam.mx/CongresoTA/memorias2024/Mem2024_Paper02.html